Inteligência artificial é capaz de predizer estruturas de proteínas com alta similaridade em relação a estruturas experimentalmente descritas, revolucionando a biotecnologia.
Descrever a estrutura de proteínas é um desafio atual da ciência. No entanto, essa predição é indispensável para entender seu funcionamento em organismos vivos e para aplicações da proteína pura em soluções farmacológicas e industriais. Atualmente as metodologias mais confiáveis para predizer a estrutura tridimensional de proteínas são a cristalografia de raio-x, a ressonância magnética nuclear e a crio-microscopia eletrônica, porém essas metodologias são de difícil execução e não são rápidas. Nesse sentido, muitos grupos têm tentado desenvolver metodologias computacionais para predição de estruturas proteicas para otimizar esse tipo de trabalho. Dentre eles o programa AlphaFold da DeepMind (rede de inteligência artificial do Google) aparece como ferramenta revolucionária para predizer essas estruturas 3D de proteínas, apresentando ótimos resultados em curtos períodos de tempo, sendo quase indistinguíveis daqueles experimentais.
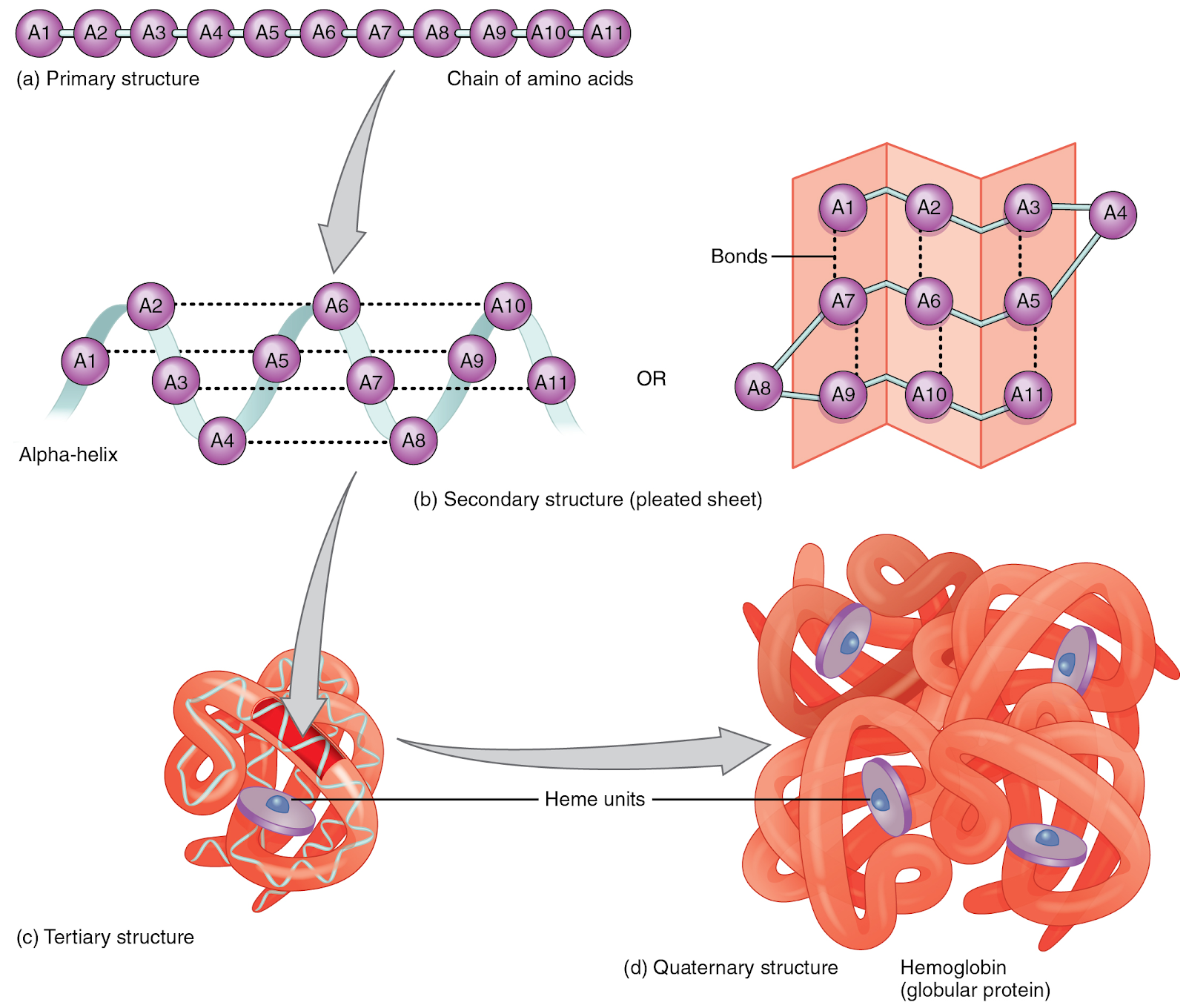
Proteínas são macromoléculas biológicas que desempenham papéis fundamentais em todos os organismos vivos, desde a coordenação de reações essenciais para manutenção da vida até a composição de estruturas biológicas. Essas moléculas são construídas a partir de informações presentes no DNA e possuem 4 níveis estruturais. O nível 1 corresponde a estrutura 1D da proteína, representada pela sequência de aminoácidos traduzida a partir do código genético. Os aminoácidos na sequência da proteína interagem entre si, inicialmente dando origem à estrutura secundária, ou estrutura 2D, adquirindo conformações chamadas, por exemplo, beta-folhas e alfa-hélices. O nível 3 é aquele em que a proteína está no nível máximo de enovelamento e se apresenta numa conformação 3D, chamada de nativa. É somente a partir dessa conformação estrutural que a proteína é capaz de desempenhar seus papéis biológicos. Sendo assim, podemos falar que entender sua estrutura 3D é essencial para descobrir sua atividade biológica e estudar aplicações biotecnológicas para essas moléculas. Já o quarto nível organizacional remete à complexos proteicos compostos pela interação de estruturas terciárias.
Muitas proteínas são utilizadas como fármacos, como é o caso da lactase (enzima usada para tratar intolerância à lactose), insulina (hormônio usado no tratamento de diabetes) e a asparaginase (usada para tratar leucemia). Por outro lado, muitas proteínas são utilizadas em diversos setores da indústria. Assim, é essencial entender sua estrutura e função para a sua aplicação biotecnológica. Além disso, experimentos para o design e prospecção de novas drogas, bem como análises de reposicionamento de fármacos, dependem muito de estruturas bem caracterizadas das proteínas-alvo.
Recentemente, esse tipo de estudo foi fundamental para elucidar proteínas que compõem o novo coronavírus causador da COVID-19, ajudando o entendimento da doença, além de gerar dados para possíveis tratamentos e formas de prevenção. Ainda em novembro desse ano, pesquisadores da UNESP conseguiram decifrar a estrutura de uma de proteína e isso permitiu a busca de fármacos para elefantíase e leishmaniose cutânea.
Dada a importância da elucidação de estruturas proteicas de modo eficiente e rápido para ciência aplicada, o AlphaFold 2 foi considerado uma revolução na 14ª edição do Experimento Comunitário de Avaliação Crítica de Técnicas para Predição de Estrutura de Proteínas, conhecido como CASP. O CASP é um concurso anual que fornece um mecanismo independente para a avaliação dos métodos de modelagem da estrutura de proteínas, ou seja, elucidação de estruturas proteicas através de métodos computacionais. Esse tipo de metodologia usa como base a sequência de aminoácidos das proteínas (sua estrutura primária) para gerar estruturas 3D, calculando através de algoritmos a posição correta dos aminoácidos na estrutura predita. Os pesquisadores ensinaram à inteligência artificial (IA) do AlphaFold a calcular essas estruturas mostrando-lhe as sequências e estruturas de cerca de 100.000 proteínas conhecidas.
Através dessa metodologia, o AlphaFold 2 foi bem mais eficiente que os demais grupos do CASP14, edição mais recente do concurso. Cerca de 100 grupos de pesquisa participaram, dentre eles pode-se citar a Microsoft e a empresa chinesa de tecnologia, Tencent, além de grupos acadêmicos. O algoritmo dessa IA apresentou uma pontuação de 90 em termos de acurácia e similaridade com estruturas de proteínas experimentalmente caracterizadas numa escala de 0 a 100, enquanto os resultados dos demais grupos giraram em torno de 75 pontos. Isso significa que o AlphaFold 2 é capaz de gerar estruturas tridimensionais de proteínas muito similares às proteínas reais, o que implica numa revolução de técnicas de predição de estruturas proteicas e na mimetização de estudos com aplicação farmacológica e demais setores da indústria que requererem essas abordagens.
News: Iasmin Taveira
Fontes e mais informações sobre o tema:
Matéria no site Nature News, intitulada “‘It will change everything’: DeepMind’s AI makes gigantic leap in solving protein structures”, publicada em 30 de novembro de 2020.
https://www.nature.com/articles/d41586-020-03348-4
Texto no site do AlphaFold/DeepMind, acesso em 03 de dezembro de 2020.
https://deepmind.com/research/case-studies/alphafold#what_is_alphafold_
Matéria do site Agência FAPESP, intitulada “Pesquisa revela estrutura de proteína e permite busca de fármacos para elefantíase e leishmaniose cutânea”, publicada em 30 de novembro de 2020.
https://agencia.fapesp.br/pesquisa-revela-estrutura-de-proteina-e-permite-busca-de-farmacos-para-elefantiase-e-leishmaniose-cutanea/34710/
(Editoração: André Pessoni)