Um novo estudo fornece fortes evidências de que as raízes genéticas do autismo também estão em regiões do genoma que não codificam proteínas.
É muito comum que muitas doenças estejam associadas a mutações em um ou vários genes. No entanto, o estudo de genomas têm revelado cada vez mais associações de doenças a regiões do DNA que não são genes, chamadas de regiões não codificantes. No caso do autismo, os cientistas agora estão mais certos que as bases genéticas para essa doenças estão não apenas nos genes, mas também nessas regiões. Estudos baseados em estatísticas sugeriram que a região não codificadora do genoma contém variantes genéticas que contribuem para o autismo, e um novo trabalho pode ser o primeiro a identificar três dessas mutações, segundo Tychele Turner, professora assistente de genética da Universidade de Washington em St. Louis, que liderou a pesquisa.
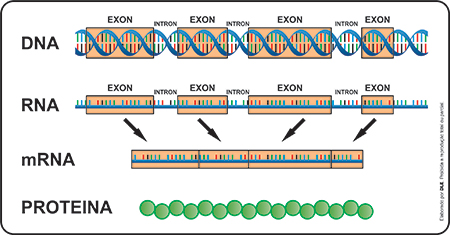
O DNA é uma molécula que contém sequências de informação que trazem códigos para a formação de moléculas de RNA que, por sua vez, repassam essa informação para uma maquinaria celular que a traduz e forma as proteínas. Então, as proteínas desenvolvem as mais diversas funções nas células, como produzir energia, carregar oxigênio e nutrientes, regular o funcionamento de órgãos, entre muitas outras. Porém, o DNA também contém muitas regiões com sequências que não trazem informação para produção de nenhuma proteína. Quando foi descoberto, esse tipo de DNA foi apelidado de DNA lixo, pois aparentemente não tinha nenhuma função. No entanto, depois de alguns anos, os cientistas passaram a ver que este apelido estava muito equivocado. O estudo do genoma humano trouxe uma informação desconcertante: apenas 2% de todos os nossos genes são codificantes, ou seja, irão resultar, após a tradução, na produção de proteínas. Esse é um número muito aquém das expectativas dos pesquisadores da época.
Hoje se sabe que, nesses 98% de DNA não-codificante, 27% são regiões de íntrons e UTRs (regiões não traduzíveis, que estão na molécula de RNA mensageiro mas que não fazem parte da tradução em proteína); 25% são regiões associadas a elementos regulatórios e a genes de RNAs não codificantes; e 46% são sequências repetitivas. Essas sequências repetitivas, por sua vez, podem atuar em papéis estruturais a nível de cromossomo. Já as regiões regulatórias que atuam na expressão gênica são, por exemplo, as regiões promotoras, que não são transcritas, mas controlam quando um gene deve ou não ser transcrito, portanto ativando ou desativando um gene. Em resumo, o DNA não-codificante não contém instruções para produção de proteínas, mas em vez disso influencia em quando, onde e como os genes são expressos. Assim, na ciência contemporânea já existem muitas pesquisas que buscam entender as funções desse tipo de DNA, e inclusive revelar suas associações com doenças.
Em seu trabalho, Turner e sua equipe pesquisaram um banco de dados de 544 sequências não codificantes do genoma que estavam ativos no cérebro de embriões de camundongos. Esses chamados segmentos enhancers, ou segmentos “intensificadores”, aumentam a transcrição de um gene. Eles então pesquisaram sequências de genoma inteiros de 2.671 famílias com uma criança autista, presentes no banco de dados Simons Simplex Collection para ver quais desses enhancers continham mutações de novo (as alterações que surgem num indivíduo sem que nenhum dos seus progenitores apresente a mesma alteração).
Um enhancer em particular, conhecido como hs737, mostrou significativamente mais mutações de novo em muitas famílias. Notavelmente, essas mutações em hs737 apareceram em três pessoas com autismo, sem outras variantes conhecidas relacionadas ao autismo. Em experimentos no laboratório com linhagens de células neuronais, as três mutações fizeram com que o enhancer diminuísse, ao contrário do esperado, a expressão do gene. Por sua vez, mapas existentes de interações do genoma em células cerebrais de camundongos e humanos mostraram que h737 regula um gene chamado EBF3.
Para a surpresa dos pesquisadores, EBF3 é um gene bem estudado. Mutações no EBF3 causam condições como hipotonia, ataxia e síndrome do desenvolvimento retardado (sigla HADDS em inglês), caracterizadas por baixo tônus muscular e problemas motores. A equipe descobriu que as pessoas autistas com mutações em hs737 também tinham problemas motores ou baixo tônus muscular. Isso acontece provavelmente porque – embora o EBF3 seja amplamente expresso pelo corpo – o enhancer hs737 está ativo no cérebro apenas durante o desenvolvimento do embrião, diz Turner. Portanto, as mutações no enhancer podem levar a características semelhantes, mas menos graves do que as mutações no próprio gene. “Isso é realmente interessante”, diz Turner. “Está começando a ficar claro por que algumas pessoas podem ter essas grandes síndromes, mas se você afetou apenas um regulador desse gene específico de um tecido, [o efeito] pode não ser tão grave, mas ainda será importante.”
Embora apenas um enhancer tenha atingido o nível de significância nominal neste estudo, com o tempo, mais intensificadores podem estar ligados ao autismo, afirmam os pesquisadores e outros cientistas. A equipe de Turner compartilhou os resultados preliminares deste trabalho na Conferência da Sociedade Americana de Genética Humana de 2020 e os publicou em 13 de julho de 2021 na revista Human Genomics. Agora, o laboratório de Turner está trabalhando no desenvolvimento de uma cepa de camundongos que não tem o enhancer hs737, a fim de entender melhor como as mutações nesse trecho de DNA não codificador afetam o comportamento.
Colaboração: Jéssica de Moura Soares sobre a autora
 Jéssica de Moura Soares é Bacharel em Biotecnologia pela Universidade Federal do Ceará (UFC), Mestre e agora doutoranda em Bioquímica pela Universidade de São Paulo (USP). Se interessa por iniciativas onde a ciência ajuda a transformar o mundo em um lugar mais sustentável e igualitário. Entusiasta da divulgação científica, acredita que a ciência tem que ser de fácil acesso a todos.
Jéssica de Moura Soares é Bacharel em Biotecnologia pela Universidade Federal do Ceará (UFC), Mestre e agora doutoranda em Bioquímica pela Universidade de São Paulo (USP). Se interessa por iniciativas onde a ciência ajuda a transformar o mundo em um lugar mais sustentável e igualitário. Entusiasta da divulgação científica, acredita que a ciência tem que ser de fácil acesso a todos.
Fontes e mais informações sobre o tema:
Matéria no site Spectrum News, intitulada “Mutations in the noncoding genome contribute to autism”, publicada em 02/08/21. https://www.spectrumnews.org/news/mutations-in-the-noncoding-genome-contribute-to-autism/
Artigo publicado no periódico Human Genomics, intitulado “Coding and noncoding variants in EBF3 are involved in HADDS and simplex autism” de autoria de Padhi, Hayeck, Cheng e colaboradores, em 13/07/2021. https://pubmed.ncbi.nlm.nih.gov/34256850/
Matéria no site Hemocentro RP (Casa da Ciência), intitulada “O lado negro do genoma”, publicado em outubro de 2015. http://www.ead.hemocentro.fmrp.usp.br/joomla/index.php/noticias/adotepauta/675-o-lado-negro-do-genoma
(Editoração: André Pessoni e Iasmin Cartaxo Taveira)

