#Mapa da Ciência (Pessoas por trás das descobertas científicas)
O papel dos RNAs nas redes de regulação dos genes
| DESTAQUES: • Arqueas são um grupo de seres vivos pouco conhecidos e estudados; • Esse grupo tem mecanismos genéticos mais similares aos nosso do que as bactérias; • O Laboratório de Biologia Sistêmica de Microorganismos estuda o papel dos RNAs de arqueas na regulação gênica; • A integração de dados em escala de “ômicas” usando bioinformática, além de técnicas de manipulação e extração de material genético de arqueas, estão entre as abordagens do Laboratório de Biologia Sistêmica de Microorganismos. |
Bactérias e fungos são os microrganismos mais conhecidos e estudados em diversos contextos, seja em relação ao ambiente, à saúde humana e animal ou na pesquisa básica. Entretanto, existe um grupo de microrganismos, conhecido como arqueas, que apresentam características bastante peculiares e ainda pouco estudadas.
Ao se observar uma arquea pelo microscópio óptico, a mesma apresenta características morfológicas de uma bactéria, devido ao seu tamanho e à ausência de núcleo e organelas na célula. Ademais, em nível de suas moléculas internas, a organização do genoma também é parecida com a das bactérias, apresentando um genoma compacto e circular. No entanto, as arqueas também apresentam características únicas, como lipídeos de membrana e células com morfologias bastante peculiares. Além disso, estudos moleculares têm revelado o papel fundamental desses microrganismos na origem dos eucariotos, revelando que seu mecanismo de expressão gênica parece uma versão simplificada da encontrada nas células eucariontes.
As arqueas estão presentes em diferentes habitats, como no solo, água, ar, e até mesmo na nossa pele, boca e sistema digestivo – mas não há relatos de nenhuma arquea considerada patogênica. As mais estudadas são as extremófilas, ou seja, as que vivem em condições ambientais extremas, seja de pH, salinidade, temperatura etc.
No Laboratório de Biologia Sistêmica de Microorganismos (LaBiSisMi) é estudada a arquea Halobacterium salinarum, um organismo halófilo, ou seja, que vive em condições extremas de sal, em concentrações de 10 a 12 a vezes a concentração de sal da água do mar. Para esse estudo são utilizadas diversas tecnologias ômicas para mapear os RNAs expressos (área chamada de Transcriptoma), as proteínas expressas (área chamada de Proteoma) e as interações entre moléculas em diferentes condições.
Assim, com esses estudos, foi possível se chegar a um modelo de regulação gênica da arquea Halobacterium salinarum, fazendo com que ela se tornasse um organismo modelo em Biologia Sistêmica, área da ciência que busca entender um organismo como um todo. Nesse contexto, o grupo de pesquisa do LaBiSisMi busca contribuir para a compreensão da Biologia Molecular de Arqueias e Regulação dos genes, focando nos RNAs.

Para realizar essas pesquisas, o laboratório trabalha com técnicas de microbiologia, cultivando H. salinarum, manipulando esse microorganismo geneticamente e avaliando suas características de crescimento e sobrevivência. Além disso, o laboratório utiliza técnicas de Biologia molecular e Bioquímica para extrair e estudar moléculas de interesse, como os RNAs e seus parceiros de interação (DNA e proteínas) e principalmente tecnologias ômicas para se estudar os RNAs.
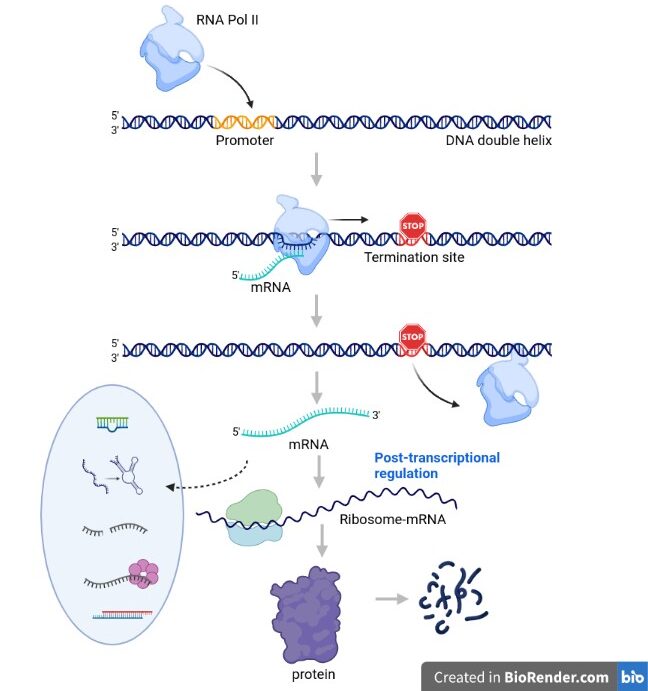
Os RNAs são moléculas fundamentais no processo de expressão gênica, seja como informação para produzir uma proteína (RNA mensageiro), como parte da maquinaria de tradução gênica (RNAs ribossomais e RNAs transportadores) e também como moléculas reguladoras ou “escultores da expressão gênica”.
O grupo de pesquisa do LaBiSisMi também tem utilizado tecnologias de sequenciamento e análise de dados a partir da bioinformática para identificar novas classes de RNAs e buscando compreender o seu papel na regulação da expressão dos genes. Diversos tipos diferentes de RNAs foram identificados pelo grupo, como RNAs circulares, RNAs relacionados com elementos móveis no genoma. E, em trabalho recente, o grupo organizou uma espécie de “atlas” em escala genômica para mapear a interação complexa entre a produção de RNAs (transcrição) e de proteínas (tradução) em H. salinarum. Dessa forma, o LaBiSisMi busca contribuir para desvendar a Biologia Molecular de arqueas e o papel dos RNAs nesse processo.
Colaboração:
 Tie Koide sobre a autora
Tie Koide sobre a autora
Tie é professora e cientista na Faculdade de Medicina de Ribeirão Preto – USP. Atua na área de Microbiologia, Bioquímica e Biologia Molecular, com ênfase em Biologia Sistêmica, analisando dados de expressão gênica em larga escala e auxiliando no desenvolvimento de ferramentas de bioinformática.
Referências:
Artigo científico intitulado “A Genome-Scale Atlas Reveals Complex Interplay of Transcription and Translation in an Archaeon” publicado na revista mSystems em 2023, de autoria de Lorenzetti, APR; Kusebauch, U; Zaramela, LS Wu, WJ; de Almeida, JPP; Turkarslan, S; de Lomana, ALG; Gomes-Filho, JV; Vêncio, RZN; Moritz, RL; Baliga NS; Koide T (https://doi.org/10.1128/msystems.00816-22)
Artigo científico intitulado “Early evolution of purple retinal pigments on Earth and implications for exoplanet biosignatures”, publicado em 2018 na revista International Journal of Astrobiology, de autoria de DasSarma, S. e Schwieterman, E.
Artigo científico intitulado “The role of predictive modelling in rationally re-engineering biological systems” publicado na revista Nature Reviews Microbiology em 2009, de autoria de Koide, T; Pang WL e Baliga NS (https://doi.org/10.1038/nrmicro2107 )
Matéria no Looking Beyond the Gene intitulada “Regulatory RNA: The sculptors of gene expression”, publicada em 23 de maio de 2019. (https://wi.mit.edu/news/regulatory-rna-sculptors-gene-expression)
Tese de doutorado defendida no programa Interunidades em Bioinformática da Universidade de São Paulo, no ano de 2019, intitulada “Regulação pós-transcricional em Halobacterium salinarum NRC-1: caracterização do gene VNG_RS05855”, de autoria de Gutierrez, J.
Vídeo “These Microbes Changed Evolutionary Biology Forever | Archaea and the Tree of Life”, de autoria do canal Science Communication Lab. (https://www.youtube.com/watch?v=rd37jBXfM4k)
Vídeo “When The Earth Was Purple”, de autoria do canal PBS Eons (https://www.youtube.com/watch?v=IIA-k_bBcL0)

- Quer ser o próximo autor colaborador do Ilha do Conhecimento? Participe!
- Não perca mais nenhuma postagem: Acompanhe nosso projeto nas redes sociais!
(Editoração: Nathália A. Khaled e Priscilla Elias F. Silva)

