#Ciência et al. (Especiais temáticos repletos de informações científicas)
O que são essas siglas e quais as diferenças entre as várias técnicas de PCR? E o que elas têm a ver com a COVID-19?
| DESTAQUES: – Siglas como PCR, qPCR, RT-PCR e similares estão presentes na mídia, mas é importante saber o que elas significam; – A técnica de PCR foi criada em 1983 e rendeu um prêmio Nobel em 1993; – Essas técnicas permitem a identificação de qualquer fragmento de material genético escolhido, a partir da produção de grandes volumes desse fragmento; – A qPCR permite a avaliação da quantidade de material genético sendo produzida em tempo real, com o uso de sondas fluorescentes; – A RT-PCR é utilizada quando o material de interesse é o RNA, e não o DNA; – Essas técnicas podem ser usadas para o diagnóstico preciso de doenças como COVID-19. |
Ultimamente, nos mais diversos meios de comunicação, popularizaram-se as discussões a respeito de técnicas e metodologias científicas utilizadas para o diagnóstico da COVID-19, como a RT-qPCR (do inglês Reverse Transcriptase Quantitative PCR), o método mais eficaz para se diagnosticar um paciente.
Esse teste ficou conhecido como “o teste do cotonete no nariz”, mas você já se perguntou como ele funciona? Como e quando surgiu essa metodologia científica tão avançada? Você sabia que existem ainda outras variantes desta técnica, chamadas de PCR, qPCR ou até mesmo RT-PCR? Mas, além das letras na sigla, quais as verdadeiras diferenças por trás de cada uma dessas técnicas?
Esse texto busca trazer essas respostas para você, que está cheio de dúvidas a respeito dessas interessantes – e extremamente versáteis – ferramentas das ciências biológicas. Essas metodologias são geralmente aplicadas na identificação de seres vivos a níveis bastante específicos, e para esclarecer melhor todas essas perguntas, vamos explicar a técnica juntamente com a sua história.
Em 1983 aconteceu uma das mais significativas descobertas do século XX: o cientista Dr. Kary B. Mullis desenvolveu a Reação em Cadeia da Polimerase, ou PCR (do nome em inglês Polymerase Chain Reaction). Essa técnica tornou possível obter muitas cópias de um mesmo fragmento de material genético, possibilitando, portanto, a obtenção de grandes quantidades de uma amostra genética específica.
As aplicações para essa técnica são extremamente diversas, podendo ser utilizada em diagnósticos de doenças, identificação de seres vivos ao nível de espécie e até mesmo como uma forma de evidência em casos policiais (área forense), visto que a técnica possibilita a produção de fragmentos do DNA de interesse partindo de quantidades de amostras muito pequenas, usando a enzima DNA polimerase, a mesma que participa da replicação do material genético nas células. Esta enzima se liga a um pequeno fragmento (o iniciador, ou primer, em inglês), desenhado especialmente para se acoplar ao DNA alvo, e produz uma sequência complementar ao fragmento de DNA de interesse, escolhido antes do início da análise.
O primeiro estudo detalhando a metodologia da técnica foi publicado no periódico científico Science, no ano de 1985, revolucionando a ciência e as possibilidades de descobertas ao se trabalhar com o DNA. Porém, essa metodologia ainda apresentava uma série de desafios, visto que é composta de 3 etapas.
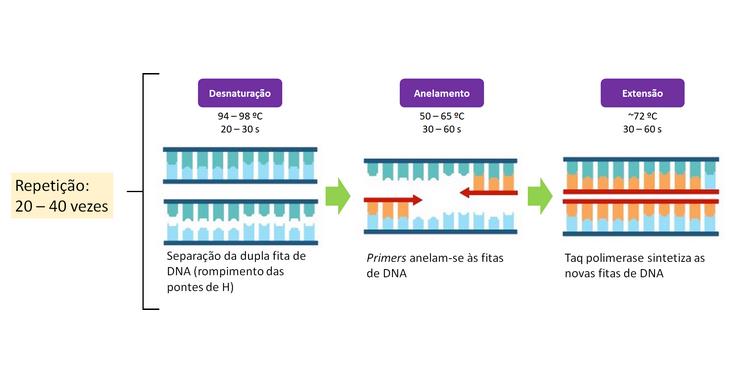
A realização de 20 a 40 ciclos promove a amplificação da região que se pretende analisar, seja ela de um gene humano específico, de microrganismos ou basicamente de qualquer material genético que se deseja multiplicar para se analisar posteriormente.
Em 1986, foi descoberta a Taq DNA polimerase, da bactéria Thermus aquaticus, um organismo adaptado a viver em temperaturas de até 100ºC. Usar a enzima dessa bactéria permitia que a reação fosse realizada sem interrupções, visto que antes dessa descoberta eram utilizadas polimerases de bactérias comuns, Escherichia coli, as quais precisavam ser adicionadas novamente a cada um dos ciclos realizados, visto que essas desnaturam junto das duplas fita de DNA no passo de desnaturação (ver figura), que eleva as amostras até 98ºC.
No ano de 1987, a técnica foi patenteada e no mesmo ano foram criados os aparelhos termocicladores, permitindo a automatização desse processo e aumentando a sua praticidade. Tamanho foi o impacto da técnica de PCR que, em 1993, o Dr. Kary B. Mullis foi premiado com o Nobel de Química por sua invenção. A partir desse ponto, começaram a surgir diversas variações da técnica e aplicações das mais diversas formas; e é nesse contexto que surge a análise tão utilizada hoje em dia nos diagnósticos de COVID-19, a RT-qPCR. Certo, mas o que significam todas essas letras adicionadas antes de PCR? Muita calma… Vamos chegar lá!
Em 1991, foi desenvolvido o método de teste TaqMan, o que marcou o início da era das PCRs Quantitativas, também conhecidas como PCR em tempo real, ou até mesmo qPCR. Porém, essa técnica começou a ser mais explorada somente a partir de 2003, com a criação de aparelhos e materiais específicos para realizar essa análise de forma mais efetiva.
Essa metodologia se parece muito com a PCR original, porém com a diferença que são adicionadas sondas fluorescentes de DNA junto das amostras, as quais emitem fluorescência a cada ciclo realizado pelo aparelho. Portanto, durante a amplificação do material genético, a sua quantidade é medida pela quantidade de fluorescência emitida pelo produto a cada ciclo. Isso é possível somente com a utilização de um sistema de equipamentos com monitoramento da fluorescência emitida, possibilitando uma quantificação mais exata de quanto material genético existia na amostra inicial, abrindo ainda mais opções e oportunidades de análises a serem feitas.
Já explicamos, portanto, sobre a PCR e a qPCR, porém de onde vem o “RT”, da RT-PCR e RT-qPCR? Essas siglas costumam confundir até mesmo pessoas experientes na área. Ao contrário do que se pensa, o RT não vem de Real Time. RT, nesse caso, diz respeito a uma metodologia com uso de uma Transcriptase Reversa (no inglês Reverse Transcriptase).
A Transcriptase Reversa é uma enzima comumente encontrada em vírus, como os retrovírus (incluindo HIV, causador de AIDS; e o vírus da hepatite B). A enzima desempenha a atividade de Transcrição Reversa, responsável pela síntese de um DNA complementar a partir de um RNA molde. Nos vírus citados, por exemplo, essa enzima é importante durante o ciclo de replicação deles. O emprego da Transcriptase Reversa na biologia molecular, incluindo a reação de RT-qPCR, é muito importante quando se deseja partir de uma amostra inicial de RNA.
Enfim, chegamos nos testes de COVID-19. Como muitos já devem saber, o SARS-CoV-2 é um vírus que tem como base de seu material genético não o DNA, mas sim o RNA. Portanto, torna-se necessário que esse material genético em forma de RNA seja transformado em DNA, antes de ser realizada a PCR em tempo real para o diagnóstico. Sem esse passo inicial realizado pela Transcriptase Reversa não haveria nenhum DNA na amostra, apenas RNA, e nada seria amplificado pela DNA polimerase, impossibilitando a detecção do vírus. Portanto, a RT-qPCR é muito importante para identificar se o Coronavírus está presente ou não e qual a quantidade inicial dele nas amostras de swab nasal e da faringe.
É válido lembrar que muitos outros testes que utilizam metodologias diferentes também são muito úteis na luta contra o Corona vírus, como os testes que procuram por anticorpos específicos para o vírus em amostras de sangue. Porém, esses testes possuem uma sensibilidade menor, e, portanto, não são tão confiáveis quanto o teste que utiliza a RT-qPCR. Além disso, é válido dizer que os testes que buscam por anticorpos são mais eficazes em mostrar pessoas que já tiveram a doença no passado, e não pessoas que estão com a doença no momento da realização do teste, o que torna os testes por RT-qPCR essenciais em estratégias públicas para detecção, tratamento e isolamento de pessoas que estão doentes e que seriam possíveis transmissores da doença.
–
Colaboração: Rafael Sanchez Luperini sobre o autor
Renato Augusto Corrêa dos Santos sobre o autor
Fontes consultadas:
– Artigo científico intitulado “A Real Time PCR strategy for the detection and quantification of Candida albicans in human blood.”, publicado na Revista do Instituto de Medicina Tropical de São Paulo em 2020, de autoria de Busser, F. e colaboradores;
– Artigo científico intitulado “A New Age in Molecular Diagnostics for Invasive Fungal Disease: Are We Ready?”, publicado na revista Frontiers in Microbiology em 2020, de autoria de Kidd, S. e colaboradores;
– Artigo científico intitulado “Development of a quantitative real-time PCR assay using SYBR Green for early detection and quantification of Austropuccinia psidii in Eucalyptus grandis.” publicado na revista European Journal of Plant Pathology em 2018, de autoria de Bini, A. e colaboradores;
– Artigo científico intitulado “Real-time PCR-based method for rapid detection of Aspergillus niger and Aspergillus welwitschiae isolated from coffee.” publicado na revista Journal of microbiological methods em 2018, de autoria de Von Hertwig, A. e colaboradores;
– Matéria no site da empresa Kasvi intitulada “História e evolução da técnica de PCR (Polymerase Chain Reaction ou Reação em Cadeia da Polimerase)” publicada em 18/06/2015. (Website);
– Matéria no site da empresa Kasvi intitulada “Qual a diferença entre PCR e qPCR?” publicada em 30/04/2015. (Website);
– Matéria no Blog Biomedicina Padrão intitulada “A evolução da PCR” publicada em 05/12/2013. (Website);
– Matéria no Blog Biomedicina Padrão intitulada “Reação em Cadeia da Polimerase – PCR” publicada em 14/06/2020. (Website).

- Quer ser o próximo autor colaborador do Ilha do Conhecimento? Participe!
- Não perca mais nenhuma postagem: Acompanhe nosso projeto nas redes sociais!
(Editoração: Beatriz Spinelli, Fernando Mecca e Caio Oliveira)

