#Ciência et al. (Conteúdos repletos de informações científicas)
Criando uma forma de diagnóstico usando o computador
| DESTAQUES: • Hanseníase é uma doença que afeta a pele e o sistema nervoso periférico; • Nosso sistema imunológico combate a bactéria por meio do reconhecimento de epítopos; • A Bioinformática nos permite descobrir esses epítopos por meio de softwares; • A partir dos epítopos descobertos, podemos criar testes diagnósticos e vacinas. |
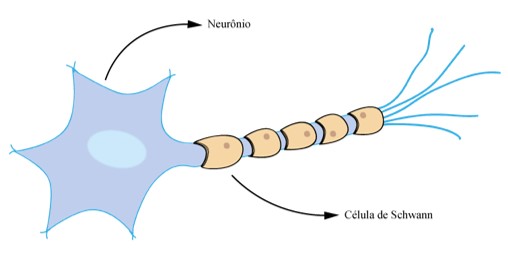
A hanseníase é uma doença causada por uma bactéria em forma de bacilo que invade as células de Schwann e gera falta de sensação tátil e térmica na região afetada da pele. Visualmente, a hanseníase se manifesta como lesões menos pigmentadas ou avermelhadas com perda de sensibilidade. Por muitos anos ela foi chamada de lepra, entretanto, esse nome abrange muitas doenças dermatológicas e ficou associado à um estigma social por causa da deformação que ela pode causar.
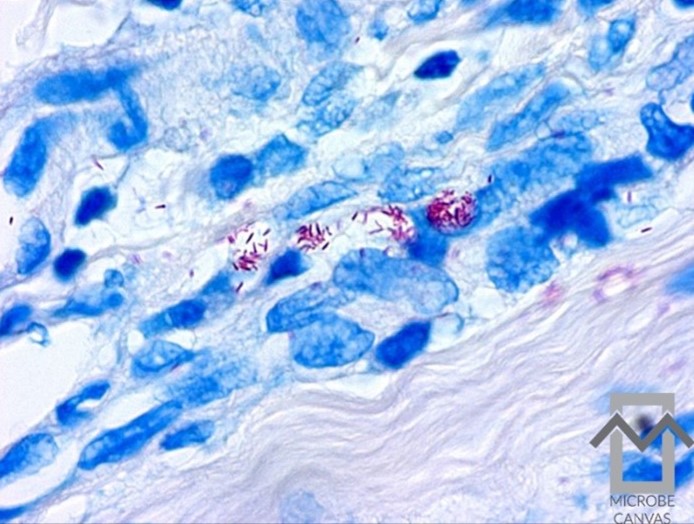
Por isso, a lepra causada pelo Bacilo de Hans ou Mycobacterium leprae e Mycobacterium lepromatosis, foi rebatizada como hanseníase ou ainda, doença de Hans. Esse nome é uma homenagem ao primeiro cientista a conseguir identificar a bactéria, Gerhard Armauer Hansen, em 1873. Mesmo sabendo qual é o agente etiológico da doença de Hans, o tratamento é muito difícil. Assim como no tratamento da tuberculose, ele leva meses de uso de antibióticos. E a perda da sensibilidade não é reparada mesmo após a cura do paciente.
A lesão do sistema nervoso periférico pode levar ainda à perda de função de membros, por exemplo: a lesão do nervo ulnar, que deixa a mão em uma posição característica que é conhecida como mão em garra.
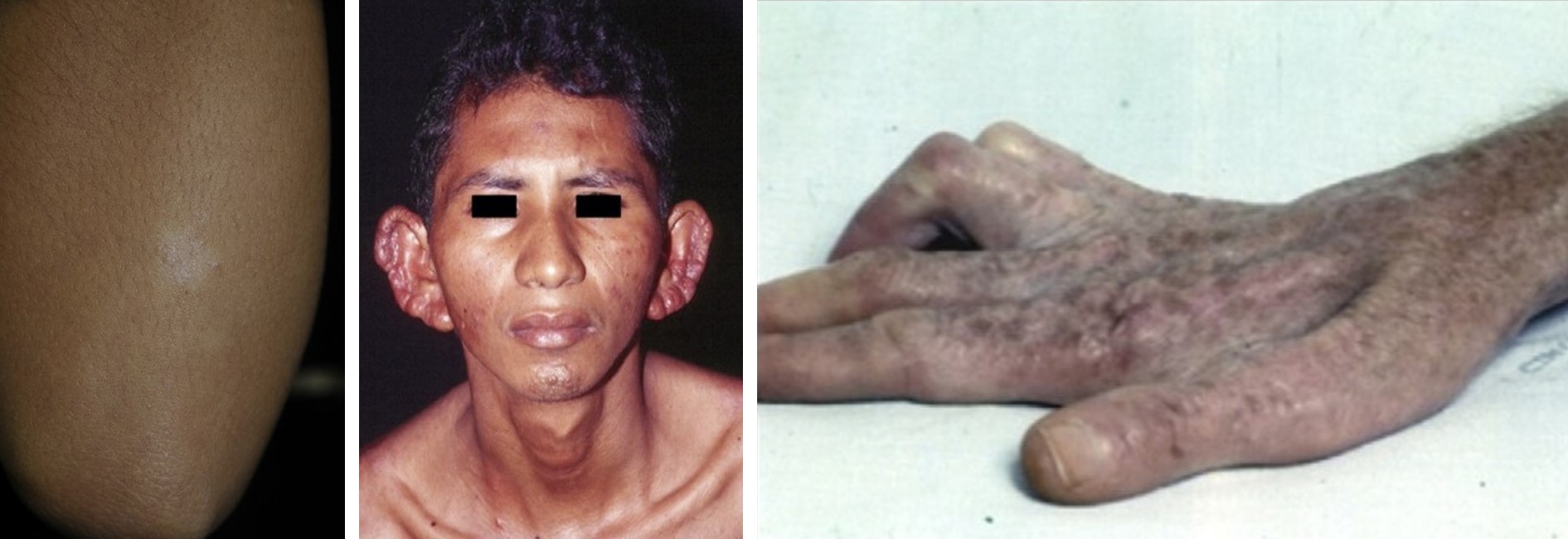
A hanseníase faz parte de um conjunto de doenças chamado de Doenças Tropicais Negligenciadas (DTN – mais informações http://ilhadoconhecimento.com.br/doencas_tropicais_negligenciadas/) juntamente com a tuberculose, leishmaniose, doença de Chagas, dengue, entre outras, que são consideradas doenças que acometem principalmente países localizados nas regiões tropicais e empobrecidos, por isso são negligenciadas.
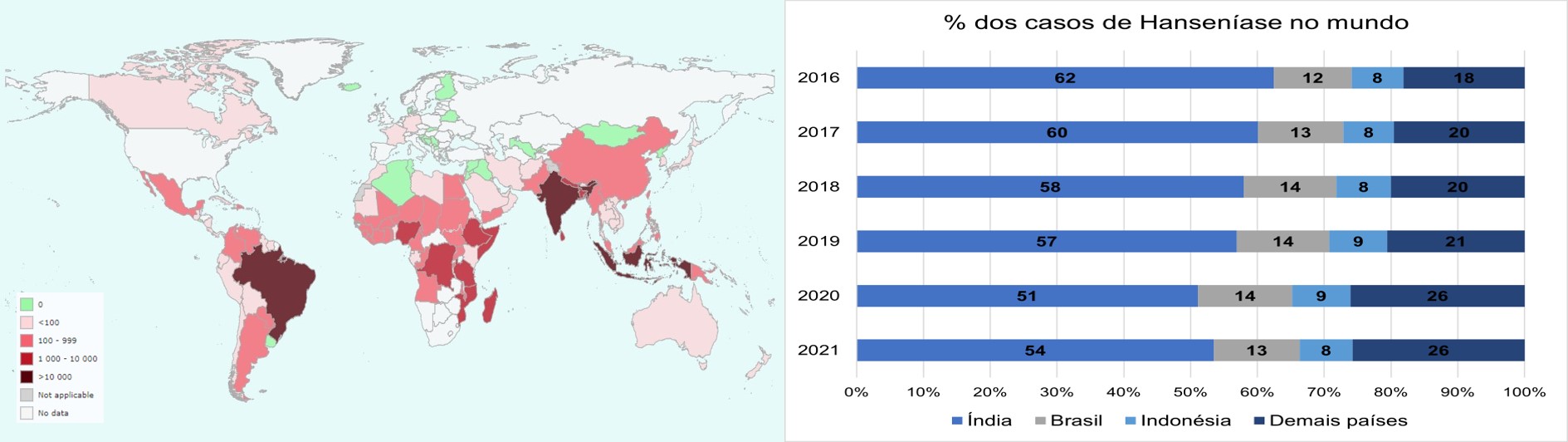
Desde 2005, Índia, Brasil e Indonésia lideram, respectivamente, o ranking de países com maior número de casos de hanseníase notificados1. Juntos, eles somam mais de 70% dos casos mundiais. Um dos maiores complicadores no combate a essa doença é a dificuldade de diagnóstico, que é feita basicamente de forma clínica, ou seja, pelo exame físico do médico, pois poucos exames laboratoriais conseguem acusar a infecção e nenhum deles consegue detectar a doença em todas as suas formas de acometimento.
O nosso sistema imunológico (SI) nos auxilia no combate a tudo que ele considera como “estranho” ao nosso corpo. É como se ele conhecesse todo o nosso corpo e quando encontra algo diferente, ele ativa diversos mecanismos que tentam eliminar isso. Mas para que o corpo “enxergue” esse material estranho, é necessário que a célula responsável por fazer essa vigilância inicial, o macrófago, ou a célula dendrítica, mostre um “pedaço” desse material para as outras células do SI, mais especificamente, aos linfócitos. Esse pedaço do material é chamado de epítopo, e é por meio dele que nosso SI reconhece e responde a diversos microrganismos (o que vai gerar a febre e a maioria dos sintomas da doença) e proteínas (quando de alimentos ou de venenos, pode causar alergias).
Muitas das bactérias que conhecemos que causam doenças são cultiváveis em laboratório, ou seja, podemos fazer com que elas se multipliquem para que possamos estudá-las. No entanto, M. leprae e M. lepromatosis em especial não podem ser cultivadas em meios de cultura; elas podem ser mantidas apenas quando há presença de outras células, o que torna o seu estudo ainda mais desafiador. Quando investigamos qual parte de uma bactéria pode ser reconhecida pelo SI, é necessário isolar essa bactéria, já que qualquer parte de outras células pode confundir o resultado; e conseguir esse isolamento ao cultivar bactérias junto de outras células é extremamente difícil.
As células do SI utilizam receptores para reconhecerem os chamados epítopos. E para que esse epítopo se ligue ao receptor é necessário que haja um complemento entre o epítopo e o receptor. Como se fosse uma chave mestra em qualquer fechadura, só que aqui a chave seria o receptor e todas as fechaduras seriam epítopos. Esses receptores mudam de acordo com a informação genética de cada um, ou seja, o nosso DNA, mas as mudanças não são muito grandes dentro de uma mesma população, portanto temos receptores diversos em populações diferentes.
Sabendo como são esses receptores, podemos prever quais são os epítopos dentro de uma determinada proteína; ou seja, quais partes de uma proteína poderão ser reconhecidas pelo SI. E sabendo quais populações possuem cada um desses receptores, podemos direcionar a escolha de epítopos para uma determinada população – informação que seria importante, por exemplo, para o desenvolvimento de vacinas. O que é muito útil, pois pode acontecer que uma parte da proteína seja epítopo para a população brasileira, mas não para a japonesa, por exemplo. Mas sabendo tudo isso, como podemos determinar quais são os epítopos em cada proteína? Essa tarefa é realizada por uma ciência bem recente, que está se desenvolvendo muito e rapidamente com a melhora da capacidade de processamento dos computadores. Ela é a bioinformática.
A bioinformática (que já foi explicada neste artigo http://ilhadoconhecimento.com.br/o-que-e-bioinformatica-e-para-que-serve/), utilizando dados provenientes do sequenciamento genético, não depende do cultivo celular que falamos mais acima, pois todos os dados sobre a bactéria já foram obtidos. Para que tenhamos informações a partir desses dados, precisamos fazer diversas análises com diferentes softwares. Ao realizar o sequenciamento da bactéria ou de qualquer outro organismo, obtemos todas as proteínas que ele pode produzir, pois elas são fruto do DNA. Com essa informação em mãos, podemos utilizar então diversos programas que comparam os receptores com as proteínas que nos parecem mais interessantes.
Com o resultado desses programas, podemos começar a filtrar quais são os melhores epítopos para podermos usar tanto como alvos vacinais quanto como ferramenta diagnóstica. Em meu trabalho, a intenção é melhorar o diagnóstico da hanseníase, que, como citado anteriormente, é difícil. Esses epítopos então são classificados por sua capacidade de serem reconhecidos por um receptor e gerar uma resposta do SI (tudo feito de forma virtual, o que chamamos de in silico). Além desses testes, eles ainda são comparados a proteínas humanas, já que não podem ser iguais a elas, senão teremos uma resposta do SI contra o próprio corpo (doença autoimune); comparados a bancos de dados que possuem diversas proteínas consideradas tóxicas ao organismo; e comparadas a outros microrganismos evolutivamente próximos deles (pois pode ocorrer reatividade cruzada).
Após selecionarmos alguns epítopos (neste trabalho foram usados 29 epítopos diferentes), nós unimos todos eles por meio do que chamamos de linkers, os quais permitem uma melhor adaptação da estrutura da proteína, já que estamos tratando de uma proteína que não é natural. Dessa união, geramos uma proteína de um tamanho médio, e aí podemos começar a analisar essa proteína como um todo. Novamente são utilizados os bancos de dados de humanos, toxicidade e microrganismos próximos, mas dessa vez na proteína inteira. Sua estrutura terciária é também observada para sabermos se o seu formato gerado por softwares é um formato possível em seres vivos (in vivo), e não só no computador (in silico). A capacidade dessa proteína em se manter inteira em altas temperaturas, em qual pH ela se modifica, se ela é hidrofílica ou hidrofóbica e o seu peso molecular são testados para podermos entender as propriedades dela. Depois de mais alguns passos mais técnicos, nós temos uma proteína pronta in silico.
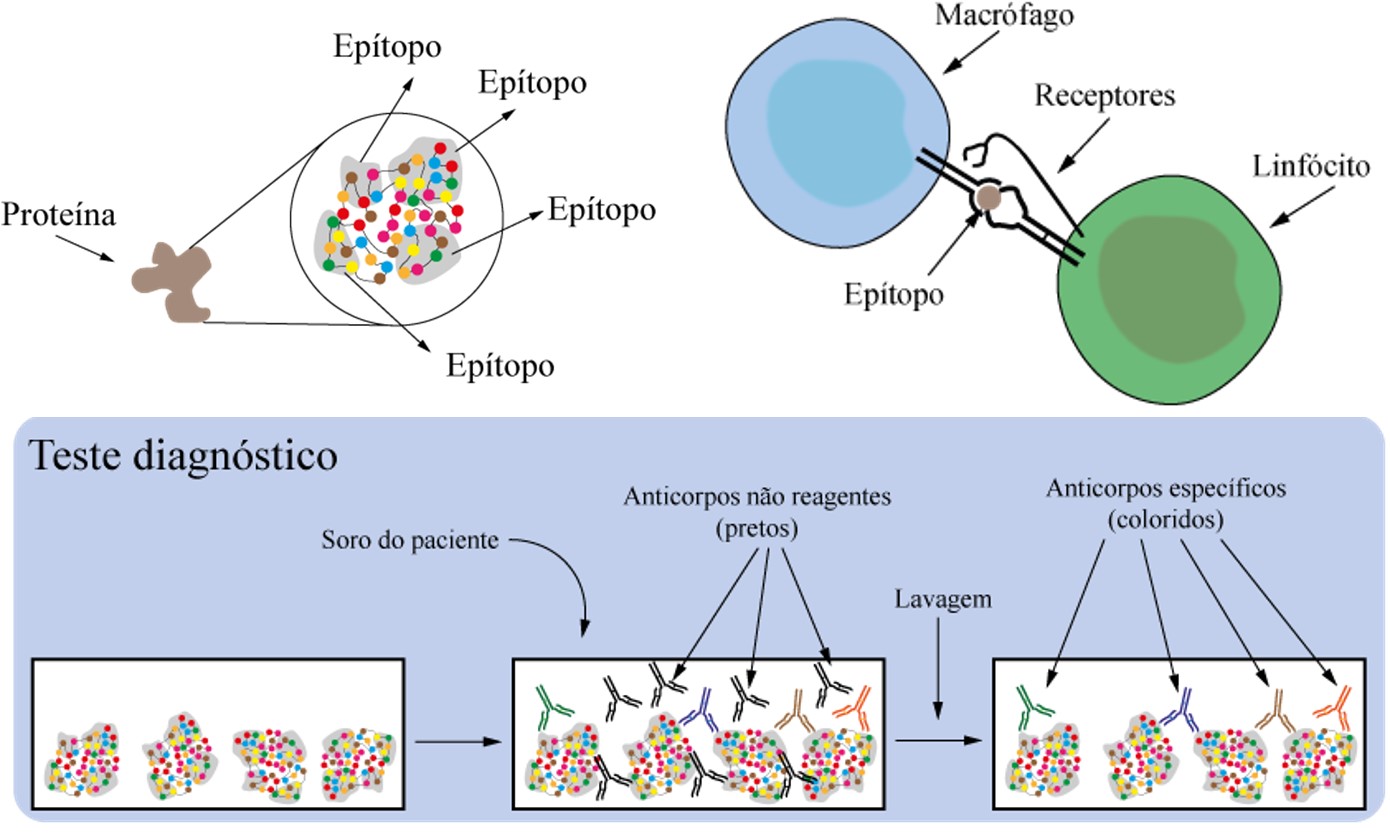
Para podermos diagnosticar qualquer doença, o nosso corpo precisa estar produzindo anticorpos, que são fruto dos linfócitos B, um tipo especial de célula que produz e libera os anticorpos. Eles são específicos para cada antígeno, quase como o receptor, mas nesse caso eles são como uma chave que abre apenas uma porta. Esses anticorpos se ligam ao microrganismo que está no nosso corpo e ajudam o SI a combatê-los. Então quando temos uma doença, esses anticorpos são liberados no nosso sangue. E coletamos o sangue com o anticorpo.
Se a nossa proteína é feita de antígenos do microrganismo que está causando a doença que, por sua vez, causou a liberação de anticorpos na nossa corrente sanguínea, esses mesmos anticorpos devem se ligar à nossa proteína. E é com essa lógica que podemos usar a proteína que nós criamos para detectar a doença. Se nada for detectado é porque a pessoa não tem anticorpos, e provavelmente, não tem a doença.
Tudo que foi citado aqui foi feito in silico (no computador). Os próximos passos desse trabalho serão produzir essa proteína de verdade e testar se os anticorpos realmente se ligam a ela e permitam que façamos o diagnóstico.
Nota:
1 Deve-se levar em conta que, justamente por serem países ou regiões empobrecidas os locais de maior incidência da doença, pode não ocorrer notificação do diagnóstico, o que limita os dados.
Colaboração:
 Marcela Rezende Lemes sobre a autora
Marcela Rezende Lemes sobre a autora
Marcela é formada em Biomedicina pela Universidade Federal do Triângulo Mineiro (UFTM), mestre em Imunologia pela mesma instituição e hoje cursa o doutorado em Bioinformática pela UFMG. Atua ainda como professora voluntária da disciplina de Bioinformática no curso de Biomedicina na UFTM. Sua maior paixão é criar figuras científicas para melhor explicar conceitos complexos de forma simples.
Referências:
Artigo científico intitulado “Analysis of antigens of Mycobacte- rium leprae by interaction to sera IgG, IgM, and IgA response to improve diagnosis of leprosy”, publicado na revista BioMed Research International em 2014, de autoria de Kumar, A., Parkash, O., Girdhar, B.
Artigo científico intitulado “Clinical aspects of leprosy”, publicado na revista Clinics in Dematoloogy em 2015, de autoria de Talhari, C., Talhari, S., e Penna, G.
Artigo científico intitulado “In silico designing of a recombinant multi-epitope antigen for leprosy diagnosis”, publicado na revista Journal of Genetic Engineering and Biotechnology em 2022, de autoria de Lemes, M., Rodrigues, T., Jaiswal, a., e colaboradores.
Artigo científico intitulado “Role of α-dystroglycan as a Schwann cell receptor for Mycobacterium leprae”, publicado na revista Science em 1979, de autoria de Rambukkana, A.
Site da World Health Organization sobre hanseníase. (Website https://www.who.int/data/gho/data/themes/topics/leprosy-hansens-disease)

- Quer ser o próximo autor colaborador do Ilha do Conhecimento? Participe!
- Não perca mais nenhuma postagem: Acompanhe nosso projeto nas redes sociais!
(Editoração: Fernando F. Mecca e Nathália Khaled)

